這裡使用 R 來分析 2015 年臺南市本土登革熱疫情狀況。
今年台灣的登革熱疫情很嚴重,台南地區的病例更突破兩萬人,以下我們利用 R 來分析台南市的登革熱疫情。
登革熱資料來源
原始資料可以從臺南市政府資料開放平台的網站上取得,以下是我整理過的資料,後續分析所需要的資料都在這裡:
- dengue-20151107-utf8.csv.zip、dengue-20151107-big5.csv.zip
- 臺南市本土登革熱病例數,截至 2015 年 11 月 7 日的資料。兩個檔案內容相同,只差在檔案編碼,一個是 UTF-8,另外一個是 BIG-5,如果是 Windows 的使用者請下載 BIG-5 的檔案,而 Mac OS X 或 Linux 則可使用 UTF-8 的檔案。
資料整理
將資料讀進 R 中,並確認資料的正確性,排除有問題的資料再進行後續分析。
將臺南市本土登革熱病例數資料讀進 R 中:
dengue <- read.csv("dengue-20151107-utf8.csv")
檢查一下資料的狀況:
str(dengue)
'data.frame': 22258 obs. of 6 variables: $ 確診日 : Factor w/ 141 levels "2015/01/06","2015/01/19",..: 1 2 3 4 5 6 7 8 9 10 ... $ 區別 : Factor w/ 41 levels "安定區","安南區",..: 12 9 6 6 6 6 6 2 6 6 ... $ 里別 : Factor w/ 530 levels " 安西里"," 仁愛里",..: 265 371 236 236 236 499 499 519 31 499 ... $ 道路名稱: Factor w/ 1489 levels ""," 建東街","?埕路",..: 764 1316 536 536 536 454 454 1341 1052 454 ... $ 緯度座標: num 23 23 23 23 23 ... $ 經度座標: num 120 120 120 120 120 ...
summary(dengue)
確診日 區別 里別
2015/09/24: 712 北區 :3529 正覺里 : 332
2015/09/18: 642 中西區 :3455 永祥里 : 319
2015/09/21: 604 永康區 :2632 大豐里 : 299
2015/09/22: 599 北 區 :2179 勝利里 : 272
2015/09/23: 584 安南區 :1831 五王里 : 269
2015/09/10: 553 南區 :1824 成德里 : 266
(Other) :18564 (Other):6808 (Other):20501
道路名稱 緯度座標 經度座標
金華路 : 690 Min. :22.63 Min. :118.8
公園路 : 671 1st Qu.:22.99 1st Qu.:120.2
西門路 : 627 Median :23.00 Median :120.2
文賢路 : 430 Mean :23.01 Mean :120.2
長榮路 : 423 3rd Qu.:23.01 3rd Qu.:120.2
府安路 : 343 Max. :24.95 Max. :121.5
(Other):19074
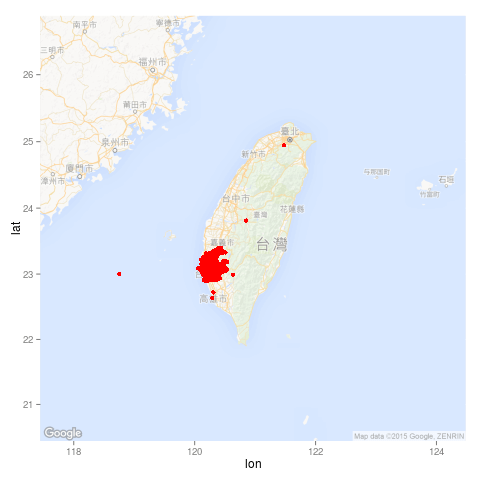
這裡的經緯度座標值有些問題,有些經緯度已經超出台南市的範圍,我們可以將資料以 ggmap 畫在地圖上來看:
library(ggmap) library(mapproj) map <- get_map(location = "Taiwan", zoom = 7, language = "zh-TW", maptype = "roadmap") ggmap(map, darken = c(0.5, "white")) + geom_point(aes(x = 經度座標, y = 緯度座標), color = "red", data = dengue)
畫出來的圖形為:
再畫一張比較大的圖:
map <- get_map(location = "Tainan", zoom = 9, language = "zh-TW", maptype = "roadmap") ggmap(map, darken = c(0.5, "white")) + geom_point(aes(x = 經度座標, y = 緯度座標), color = "red", data = dengue)
畫這張圖的時候,出現一行警告訊息,表示有三筆資料沒有畫在上面(這三筆可以從上面那張圖看出來)。
警告訊息: Removed 3 rows containing missing values (geom_point).
這是畫出來的圖形:
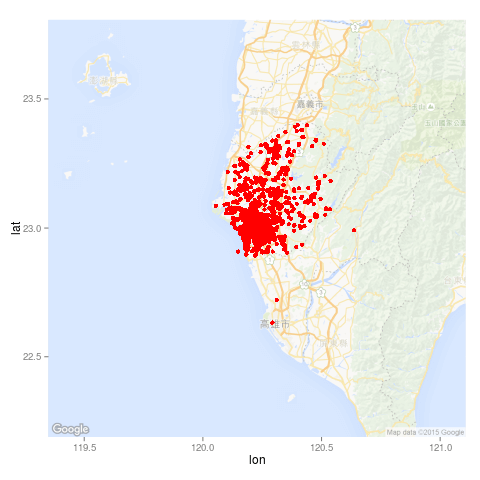
我們透過圖形來訂出一個篩選資料的方形區域:
map <- get_map(location = "Tainan", zoom = 9, language = "zh-TW", maptype = "roadmap") ggmap(map, darken = c(0.5, "white")) + geom_point(aes(x = 經度座標, y = 緯度座標), color = "red", data = dengue) + geom_rect(aes(xmin = 120, xmax = 120.6, ymin = 22.8, ymax = 23.5), alpha = 0.1)
畫出來的圖形為:
接著把實際的資料篩選出來:
filter.idx1 <- dengue$緯度座標 > 22.8 & dengue$緯度座標 < 23.5 filter.idx2 <- dengue$經度座標 > 120 & dengue$經度座標 < 120.6 dengue.tn <- dengue[filter.idx1 & filter.idx2, ]
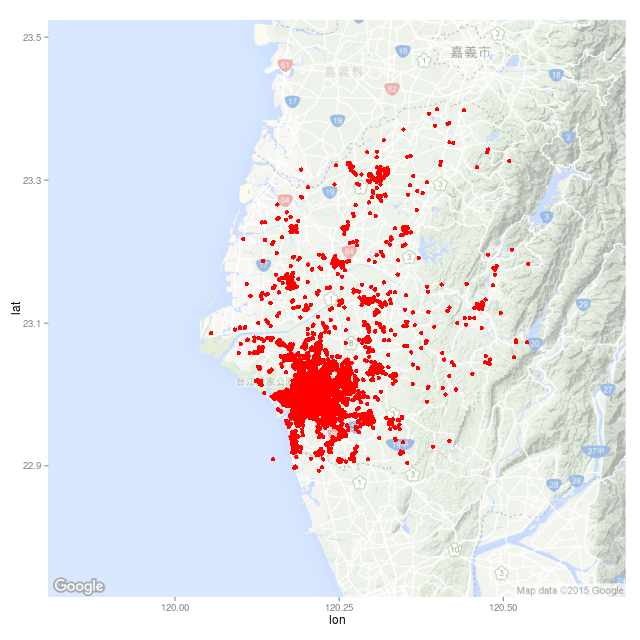
把篩選好的資料畫在地圖上:
map <- get_map(location = c(lon = 120.246100, lat = 23.121198), zoom = 10, language = "zh-TW") ggmap(map, darken = c(0.5, "white")) + geom_point(aes(x = 經度座標, y = 緯度座標), color = "red", data = dengue.tn)
畫出的圖形為
這張圖就是臺南市全年的登革熱病例分佈地圖。
從上面 dengue 的 summary 輸出中可以看到行政區的名稱有一些問題,我們將 dengue.tn$區別 的 levels 列出來看一下:
levels(dengue.tn$區別)
[1] "安定區" "安南區" "安平區" "白河區" "北 區" [6] "北區" "大內區" "東 區" "東區" "東山區" [11] "官田區" "關廟區" "歸仁區" "後壁區" "佳里區" [16] "將軍區" "柳營區" "六甲區" "龍崎區" "麻豆區" [21] "南 區" "南 區" "南化區" "南區" "楠西區" [26] "七股區" "仁德區" "山上區" "善化區" "西港區" [31] "下營區" "新化區" "新市區" "新營區" "學甲區" [36] "鹽水區" "永康區" "永康區 " "玉井區" "中西區" [41] "左鎮區"
部份的區別名稱有包含空白,而有些卻沒有,區別名稱不統一會造成程式將一個區域誤判為多個區域。這裡我們修正區別的名稱,統一將所有的空白去掉:
dengue.tn[dengue.tn$區別 == "北 區", ]$區別 <- "北區" dengue.tn[dengue.tn$區別 == "東 區", ]$區別 <- "東區" dengue.tn[dengue.tn$區別 == "南 區" | dengue.tn$區別 == "南 區", ]$區別 <- "南區" dengue.tn[dengue.tn$區別 == "永康區 ", ]$區別 <- "永康區"
重新建立一次 factor,這樣可以將空的 levels 去掉:
dengue.tn$區別 <- factor(dengue.tn$區別)
然後再確認一次區別名稱:
levels(dengue.tn$區別)
[1] "安定區" "安南區" "安平區" "白河區" "北區" [6] "大內區" "東區" "東山區" "官田區" "關廟區" [11] "歸仁區" "後壁區" "佳里區" "將軍區" "柳營區" [16] "六甲區" "龍崎區" "麻豆區" "南化區" "南區" [21] "楠西區" "七股區" "仁德區" "山上區" "善化區" [26] "西港區" "下營區" "新化區" "新市區" "新營區" [31] "學甲區" "鹽水區" "永康區" "玉井區" "中西區" [36] "左鎮區"
整體趨勢分析
分析全臺南市本土登革熱病例數整體分佈與趨勢。找出疫情最嚴重的時段。
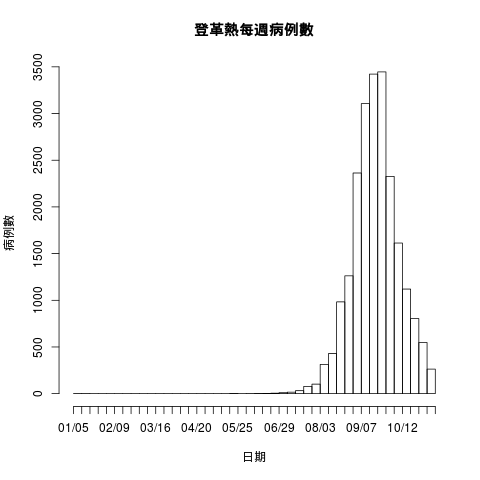
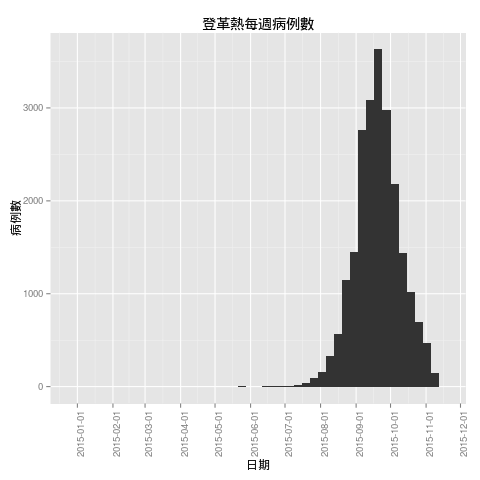
畫出每週登革熱的病例數統計圖:
hist(as.Date(dengue.tn$確診日), breaks = "weeks", freq = TRUE, main = "登革熱每週病例數", xlab = "日期", ylab = "病例數", format = "%m/%d")
畫出的圖形為
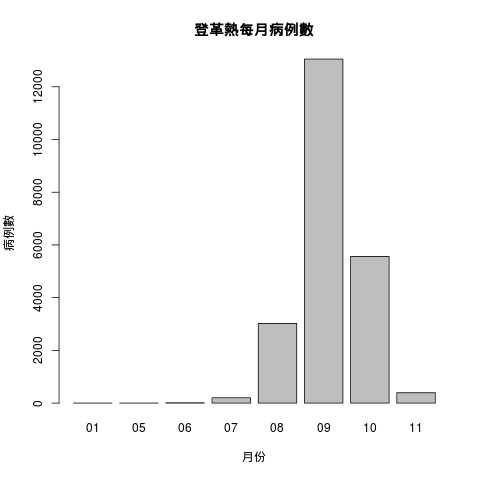
計算每個月的登革熱病例數:
dengue.tn$month <- format(as.Date(dengue.tn$確診日), "%m") table(dengue.tn$month)
01 05 06 07 08 09 10 11
2 2 11 202 3023 13054 5565 393
畫圖:
barplot(table(dengue.tn$month), xlab = "月份", ylab = "病例數", main = "登革熱每月病例數")
也可以使用 ggplot2 來畫:
library(ggplot2) library(scales) ggplot(dengue.tn, aes(x=as.Date(確診日))) + stat_bin(binwidth=7, position="identity") + scale_x_date(breaks=date_breaks(width="1 month")) + theme(axis.text.x = element_text(angle=90)) + xlab("日期") + ylab("病例數") + ggtitle("登革熱每週病例數")
畫出的圖形為
從圖形上可以看出登革熱的疫情最嚴重的時期是在九月份前後。
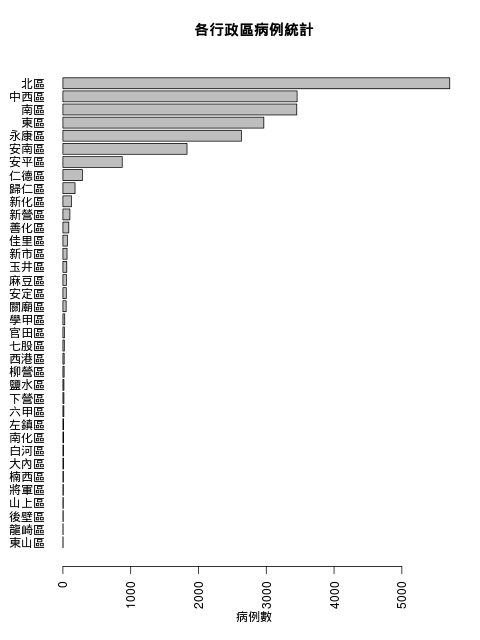
接下來分析疫情最嚴重的區域。計算各個行政區的病例總數:
dengue.region.summary <- sort(summary(dengue.tn$區別), decreasing = FALSE) dengue.region.summary
東山區 龍崎區 後壁區 山上區 將軍區 楠西區
3 3 5 6 7 8
白河區 大內區 南化區 左鎮區 六甲區 下營區
9 9 10 10 14 15
鹽水區 柳營區 西港區 七股區 官田區 學甲區
15 18 19 21 22 27
關廟區 安定區 麻豆區 玉井區 新市區 佳里區
48 50 51 57 61 67
善化區 新營區 新化區 歸仁區 仁德區 安平區
87 103 126 179 287 875
安南區 永康區 東區 南區 中西區 北區
1831 2633 2964 3450 3455 5707
畫圖:
barplot(dengue.region.summary, las = 2, horiz = TRUE, main = "各行政區病例統計", xlab = "病例數")
畫出的圖形為
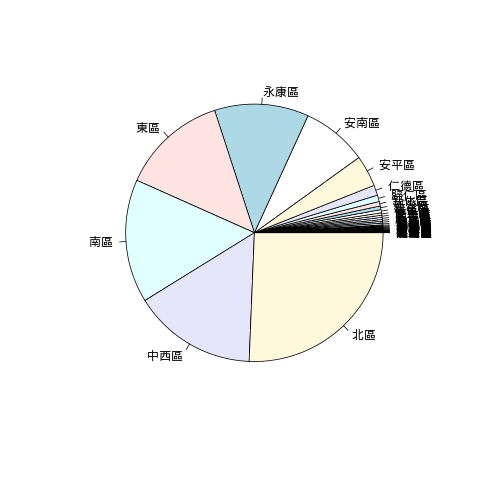
使用圓餅圖:
pie(dengue.region.summary)
畫出的圖形為
疫情最嚴重的五個區域依序為北區、中西區、南區、東區與永康區。
繼續閱讀: 12



Tim
謝謝分享
Yen
你好,
我從台南市政府下載的104年登革熱病例數資料,從20463筆開始都是亂碼,用excel讀取csv,更改任何編碼模式都無法正常顯示
程式新手用mac還沒摸熟怎麼使用emacs編輯csv檔案
想向您請教解決方式
Douglas
> dengue <- read.csv("dengue-20151107-big5.csv")
Error in file(file, "rt") : cannot open the connection
In addition: Warning message:
In file(file, "rt") :
cannot open file 'dengue-20151107-big5.csv': No such file or directory
讀不到不知道為什麼